
Les grands fleuves comme le Rhône sont des écosystèmes vitaux, mais le suivi de leurs populations de poissons est coûteuse et complexe.
Les méthodes traditionnelles telles que la pêche électrique présentent des limites, tandis que l’ADNe permet une couverture beaucoup plus étendue.
Dans cette étude, l’analyse des traces génétiques dans des échantillons d’eau collectés le long de 500 km du Rhône a permis de capturer précisément les assemblages de poissons, reflétant à la fois la présence des espèces et leur abondance relative.
Pourquoi est-ce important ?
Les écosystèmes aquatiques, en particulier les fleuves et les rivières, subissent une dégradation importante tant sur le plan de la diversité des espèces que du fonctionnement écologique. Le taux de déclin des populations aquatiques est près de deux fois supérieur à celui observé chez les espèces terrestres. Face à cette situation alarmante, il devient essentiel d’amplifier l’échelle et la fréquence des suivis de la biodiversité aquatique.
Cette étude montre également que, comparé aux méthodes traditionnelles d’échantillonnage à l’échelle locale (quelques kilomètres), le métabarcoding de l’ADNe permet d’obtenir une liste d’espèces plus exhaustive. Il est ainsi reconnu comme une méthode plus sensible que la pêche électrique ou la pêche au filet.
Les résultats ont été publiés et sont accessibles ici.
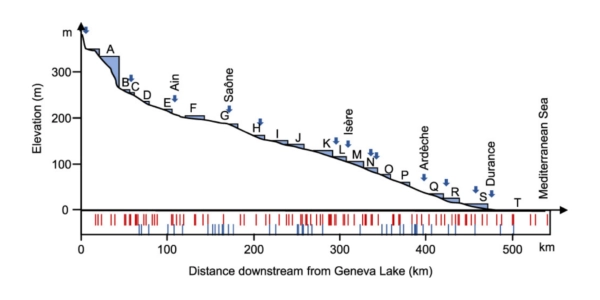
Localisation : Fleuve Rhône, du lac Léman à la mer Méditerranée (540 km).
Écosystème : Rivière.
Profondeur : Échantillonnage en surface.
Méthode d’échantillonnage : deux filtres de 30 litres sur 20 sections successives du Rhône, utilisant deux pompes péristaltiques placées de chaque côté d’un bateau.
Groupe taxonomique : poissons, avec amorces teleo.

