
La technologie ADNe offre une méthode non invasive, reproductible et indépendante de l’observateur pour le suivi de la biodiversité.
Cependant, le terme « ADNe » désigne aujourd’hui une multitude de protocoles très variables dans leur degré de sophistication — ce qui soulève une question essentielle : sont-ils tous aussi efficaces les uns que les autres ?
Pour investiguer cela, nous avons comparé les résultats obtenus avec le protocole standard de SPYGEN pour les milieux d’eau douce (30 L d’eau filtrée, 12 réplicats PCR, 500 000 lectures) à ceux issus d’une simulation basée sur un protocole basique (2 L, 8 réplicats PCR, 100 000 lectures) pour un fleuve allemand. Résultat :
- 33 % des espèces n’ont pas été détectées et
- 67 % des espèces menacées ont été manquées par le protocole basique.
Pourquoi est-ce important ?
Des technologies ADNe rigoureuses sont essentielles. Des méthodes inadéquates peuvent manquer les espèces rares (souvent plus importantes pour la conservation ou la recherche), et produire ainsi des données biaisées, entraînant des conclusions erronées.
3 éléments clés pour comparer des protocoles ADNe:
- La stratégie d’échantillonnage compte : l’ADNe étant localisé, le fait de filtrer de plus grands volumes d’eau le long de transects intégrés (plutôt qu’en points fixes) permet de capter davantage d’ADNe et ainsi d’obtenir une image plus représentative de la biodiversité d’un site.
- Les réplicats PCR augmentent la probabilité de détection : utiliser 12 réplictas PCR revient à amplifier l’ADN dans 12 sous-échantillons indépendants, ce qui améliore fortement les taux de détection et réduit les faux négatifs, en particulier avec nos amorces à haute performance.
- La profondeur de séquençage détermine la puissance de détection : plus on séquence de brins d’ADN, plus on augmente les chances de détecter des espèces. Analyser 500 000 séquences permet de mieux détecter les espèces rares ou peu abondantes — jusqu’à 5 fois mieux qu’avec les approches standards à 100 000 séquences.
Découvrez les résultats ci-dessous:
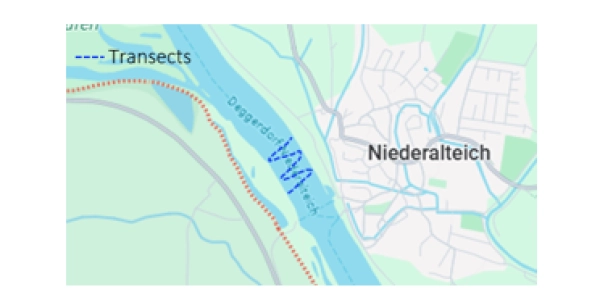
Localisation : Niederaltaich (Allemagne).
Écosystème : Fleuve Danube.
Méthode d’échantillonnage : filtration de 30 L d’eau douce à l’aide des filtres haute capacité de SPYGEN depuis un bateau naviguant d’une rive à l’autre.
Groupe taxonomique : poissons, via l’amorce teleo développée par SPYGEN.
Espèces et lectures de séquençage détectées : voir ci-dessous.
| Nom Commun | Nom Scientifique | Statut UICN | Protocole SPYGEN Volume d’eau filtré = 30 L Nombre de réplicats PCR = 12 Prof de séquençage = 500 000 |
Protocole Simplifié Volume d’eau filtré = 2 L Nombre de réplicats PCR = 8 Prof de séquençage = 100 000 |
|---|---|---|---|---|
| Brème commune | Abramis brama | LC | 50 422 | 10 084 |
| Ablette | Alburnus alburnus | LC | 36 585 | 7 317 |
| Aspe | Aspius aspius | LC | 2 132 | 426 |
| Barbeau fluviatile | Barbus barbus | LC | 63 428 | 12 686 |
| Vandoise de Vimba | Bsapa_Bbjoernkna_Vvimba | LC | 3 991 | 798 |
| Goujon à nageoires blanches | Ggobio_Ralbipinnatus | LC | 3 848 | 770 |
| Ide mélanote / Vandoise / Ablette | Lidus_Lleuciscus_Pcultratus | LC | 3 755 | 751 |
| Gardon | Rutilus rutilus | LC | 13 944 | 2 789 |
| Gardon du Danube | Rutilus virgo | LC | 6 417 | 1 283 |
| Chevaine | Squalius cephalus | LC | 2 694 | 539 |
| Perche commune | Perca fluviatilis | LC | 1 365 | 273 |
| Spirlin / Vandoise méridionale | Alburnoides bipunctatus | LC | 698 | Non Détecté |
| Goujon du Danube | Romanogobio uranoscopus | LC | 23 | Non Détecté |
| Loche franche | Barbatula barbatula | LC | 155 | Non Détecté |
| Grémille | Gymnocephalus sp. | LC | 872 | Non Détecté |
| Sandre | Sander sp. | LC | 328 | Non Détecté |
| Zingel streber | Zingel streber | LC | 364 | Non Détecté |
| Truite fario / Truite commune | Salmo trutta | LC | 287 | Non Détecté |
| Perche soleil | Lepomis gibbosus | INVASIVE | 2 266 | 453 |
| Gobie coureur | Babka gymnotrachelus | INVASIVE | 1 891 | 378 |
| Gobie à taches noires | Neogobius melanostomus | INVASIVE | 8 868 | 1 774 |
| Gobie à grosse tête | Ponticola kessleri | INVASIVE | 4 827 | 965 |
| Gobie à museau tubulaire | Proterorhinus semilunaris | INVASIVE | 2 632 | 526 |
| Nase / Souffia | Cnasus_Tsouffia | NT/LC | 7 705 | 1 541 |
| Gardon italien / Gardon de Pigus | Rutilus pigus | VU | 5 360 | 1 072 |
| Sterlet | Acipenser ruthenus | EN | 22 | Non Détecté |
| Anguille européenne | Anguilla anguilla | CR | 52 | Non Détecté |
Cette comparaison repose sur une simulation basée sur une modélisation linéaire d’un protocole eDNA simplifié, et est destinée uniquement à des fins illustratives. Elle ne reflète en aucun cas les protocoles validés d’un prestataire spécifique. L’objectif est de montrer comment certains paramètres méthodologiques clés — tels que le volume d’eau filtré, le nombre de réplicats PCR et la profondeur de séquençage — influencent la puissance de détection, et à quel point il est facile de passer à côté de certaines espèces, y compris des espèces menacées, lorsque les protocoles sont sous-dimensionnés.
Le modèle évalue la perte attendue de détection d’espèces en raison de réductions successives de : (i) volume d’eau filtré, de 30 L à 2 L (×15), (ii) nombre de réplicats PCR, de 12 à 8 (×1,5), et (iii) profondeur de séquençage, de 500 000 à 100 000 lectures (×5), entraînant une baisse globale du signal eDNA exploitable. La perte d’espèces est estimée sur la base de la probabilité de passer sous un seuil de détection de 10 lectures uniques par espèce, afin de limiter les faux positifs. Il s’agit d’un exercice théorique visant uniquement à illustrer des considérations méthodologiques générales, sans intention d’évaluer, de critiquer ou de comparer les performances d’un quelconque prestataire commercial.
Le protocole standard de Spygen pour les environnements marins et d’eau douce prévoit la filtration de 30 litres d’eau par filtre, avec 2 filtres utilisés sur le même site (soit 60 litres filtrés dans le même transect intégratif). L’expérience empirique indique que cette approche renforce encore la puissance de détection.

